Bemutatkozás, fő kutatási terület
A Fehérje Bioinformatika Kutatócsoport a Természettudományi Kutatóközpont Molekuláris Élettani Intézetében működik, vezetője Dr. Tusnády Gábor. A csoport 2019-ben a Fehérjeszerkezet Kutatócsoport (vezetője Prof. Dr. Simon István) és a Membránfehérje Bioinformatika Kutatócsoport összevonásával alakult meg.
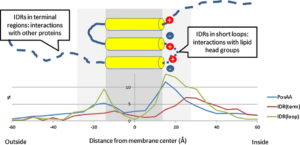
A csoport fő célkitűzése a membrán fehérjék topológiájának és 3Ds szerkezetének vizsgálata, olyan modern bioinformatika eljárások kifejlesztésével, mint a CCTOP, vagy TMFoldRec eljárások, valamint adatbázisok létrehozásával és karbantartásával, amelyek a transzmembrán fehérjék topológiájával és szerkezetével kapcsolatosak. A csoportban vizsgálják még a transzmembrán és globuláris fehérjékben előforduló rendezetlen régiókat valamint ezen régióknak a fehérje-fehérje kölcsönhatásokban betöltött szerepét és a transzmembrán fehérjék kölcsönhatási hálózatát. A bioinformatikai eljárások mellett a csoport részt vesz transzmembrán fehérjék szerkezeti és localizációs adatainak generálására alkalmas nagy áteresztő képességű módszer fejlesztésében is.
A transzmembrán fehérjék vizsgálatában elért eredményeken kívül a csoport bekapcsolódott számos új generációs szekvenáláson (NGS) alapuló kutatásba, kezdve a csirke DT40 bursal limfóma sejtvonal és a Micoplazma sp HU2014 genom szekvenálástól az általános kemoterápiás szerek mutagén hatásának és a formalinnal fixált, parafinba ágyazott tumor minták TruSeq Custom Amplicon esszé klinikai validációján át, ismert és ismeretlen virális patogének növényekben való azonosításáig.
Aktuális együttműködések
Külföldi:
- Pediatrics, Harvard Medical School; Pediatrics, Boston Children’s Hospital
Hazai:
- Onkológiai Intézet, Sebészi és Molekuláris Daganatpatológiai Centrum
- Szegedi Tudományegyetem, Orvos Vegytani Intézet
- Természettudományi Kutatóközpont, Szerves Kémiai Intézet
Főbb műszerek, berendezések
- Supermicro kompjuter klaszter (1240 CPU mag, 2.5Tb RAM, 240 Tb redundáns winchester kapacitás.
- Web szerver (40 CPU mag, 160Gb RAM, 12Tb redundáns Sata disk, 2x4Tb PCIexpress SSD disk)
- Web szerver (48 CPU mag, 390Gb RAM, 33Tb redundáns Sata disk)
- 60Tb QNAP NAS szerver
Elmúlt 10 évben végzett PhD hallgatók
Az elmúlt 5 évben megjelent legjobb 10 publikáció
- Dobson, L, Gerdán, C, Tusnády, S, Szekeres, LI, Kuffa, KR, Langó, T, Zeke, A and Tusnády, GE (2024) UniTmp: unified resources for transmembrane proteins. Nucleic Acids Res 52: D572-D578.
- Dobson, L, Szekeres, LI, Gerdán, C, Langó, T, Zeke, A and Tusnády, GE (2023) TmAlphaFold database: membrane localization and evaluation of AlphaFold2 predicted alpha-helical transmembrane protein structures. Nucleic Acids Res 51: D517-D522.
- Jambrich, MA, Tusnady, GE and Dobson, L (2023) How AlphaFold2 shaped the structural coverage of the human transmembrane proteome. Sci Rep 13: 20283.
- Zeke, A, Takács, T, Sok, P, Németh, K, Kirsch, K, Egri, P, Póti, ÁL, Bento, I, Tusnády, GE and Reményi, A (2022) Structural insights into the pSer/pThr dependent regulation of the SHP2 tyrosine phosphatase in insulin and CD28 signaling. Nat Commun 13: 5439.
- Langó, T, Kuffa, KR, Tóth, G, Turiák, L, Drahos, L and Tusnády, GE (2022) Comprehensive Discovery of the Accessible Primary Amino Group-Containing Segments from Cell Surface Proteins by Fine-Tuning a High-Throughput Biotinylation Method. Int J Mol Sci 24: 273.
- Zeke, A, Dobson, L, Szekeres, LI, Langó, T and Tusnády, GE (2021) PolarProtDb: A Database of Transmembrane and Secreted Proteins showing Apical-Basal Polarity. J Mol Biol 433: 166705.
- Dobson, L, Zeke, A and Tusnády, GE (2021) PolarProtPred: predicting apical and basolateral localization of transmembrane proteins using putative short linear motifs and deep learning. Bioinformatics 37: 4328-4335.
- Dobson, L and Tusnády, GE (2021) MemDis: Predicting Disordered Regions in Transmembrane Proteins. Int. J. Mol. Sci. 22: 12270.
- Langó, T, Pataki, ZG, Turiák, L, Ács, A, Varga, JK, Várady, G, Kucsma, N, Drahos, L and Tusnády, GE (2020) Partial proteolysis improves the identification of the extracellular segments of transmembrane proteins by surface biotinylation. Sci Rep 10: 8880.
- Müller, A, Langó, T, Turiák, L, Ács, A, Várady, G, Kucsma, N, Drahos, L and Tusnády, GE (2019) Covalently modified carboxyl side chains on cell surface leads to a novel method toward topology analysis of transmembrane proteins. Sci Rep 9: 15729.