Jelátviteli és Funkcionális Genomika Kutatócsoport
Általános kutatási terület
A Jelátviteli és Funkcionális Genomika Kutatócsoporton belül két kutatólaboratórium működik, amelyek több területen is összehangolják tevékenységüket. A Jelátviteli Laboratórium (vezetője: Buday László) elsősorban a növekedési faktorok jelátviteli pályáit vizsgálja, melyek a tirozin kináz enzimek segítségével szabályozzák a sejtek osztódását, illetve mozgását. A Funkcionális Genomika Laboratórium (vezetője: Patthy László) részben a bioinformatika, azon belül is a genom annotáció területén tevékenykedik. Ugyancsak fontos kutatási területe az orvos biológiai szempontból fontos fehérjék szerkezetének és funkciójának kísérletes vizsgálata. Mindkét Kutatólaboratórium részt vesz a Fehérjetudomány és alkalmazásai Nemzeti Programban (HunProtEx, 2018-1.2.1-NKP-2018-00005), illetve az ELTE által vezetett Felsőoktatási és Ipari Együttműködési Központ (FIEK_16-1-2016-0005) működésében.
Főbb kutatási témák összefoglalója
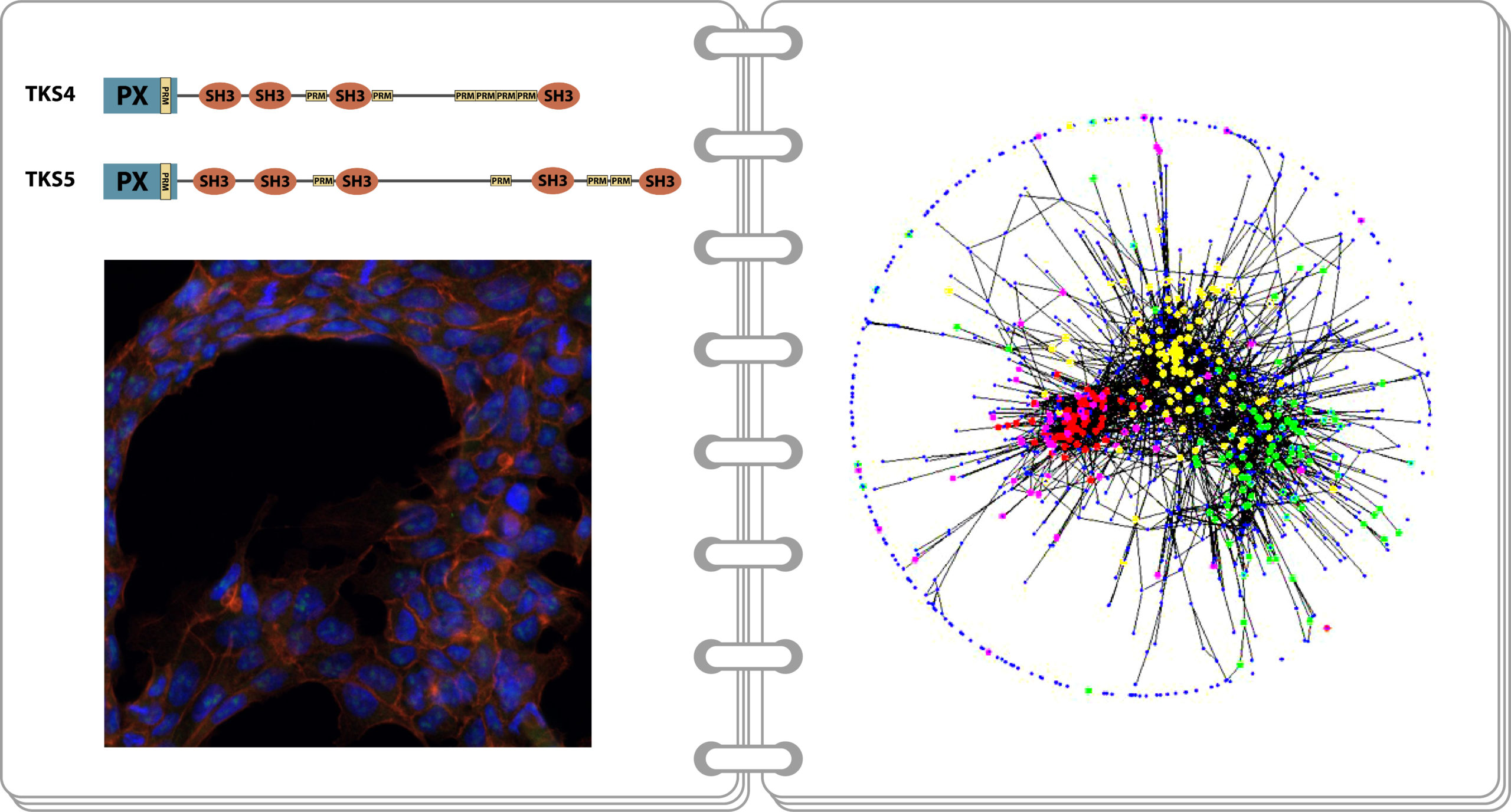
- A Tks fehérjék vizsgálata
A tirozin kináz szubsztrát (Tks) fehérjecsalád két tagja az N-terminálisan megtalálható PX domén mellett négy (Tks4) vagy öt (Tks5) SH3 domént tartalmaz. Ez a domén-szerkezet arra utal, hogy ezek a fehérjék lipid- és fehérjepartnereket képesek megkötni, ami az állványfehérjékre jellemző. A Tks4 fehérje, illetve génjének (SH3PXD2B) közvetlen orvosi jelentőségét az adja, hogy az SH3PXD2B gén mutációi okozzák a súlyos fejlődési rendellenességeket mutató emberi Frank-ter Haar szindrómát. A csoport kimutatta, hogy a Tks4 és a Tks5 fehérje egyaránt részt vesz az EGF jelpályában [1-3]. Sikerült bizonyítaniuk, hogy a Frank-ter Haar szindrómában megtalálható, a PX domént érintő pont mutáció következtében a Tks4 fehérje szerkezete károsodik és a sérült fehérje a sejtek aggreszómáiban halmozódik fel [4]. Az általuk előállított Tks4 génhiányos egérvonal felhasználásával bizonyították, hogy az Tks4-/- egerekben sérül a csontvelőből származó mesenchymális őssejtek diffrenciációja zsír-, illetve csontszövet irányában, ami részben magyarázhatja a betegekben található elváltozásokat [5]. Az elmúlt években csoportunk eddig még nem ismert funkcióit azonosította a Tks4-nek a felnőtt zsírszövet homeosztázisában [6] és a csontritkulás kialakulásában [7]. A Tks4 fehérje molekuláris működését vizsgálva megállapítottuák, hogy EGF-fel kezelt sejtekben SH2- és SH3 doméneken keresztül kötődik az őt foszforilálni képes Src tirozin kinázhoz [8].
Eddigi Tks fehérjékkel kapcsolatos munkánkat és az irodalom áttekintését egy összefoglaló cikk formájában is közöltük [9].

[1] Lányi A, Baráth M, Péterfi Z, Bögel G, Orient A, Simon T, Petrovszki E, Kis- Ttoth K, Sirokmány G, Rajnavölgyi É, Terhorst C, Buday L and Geiszt M. (2011) The homolog of the Five SH3-Domain Protein (HOFI/SH3PXD2B) Regulates Lamellipodia Formation and Cell Spreading. PLoS ONE 6, e23653
[2] Bögel G, Gujdár A, Geiszt M, Lányi Á, Fekete A, Sipeki S, Downward J and Buday L. (2012) Frank-ter Haar syndrome protein Tks4 regulates epidermal growth factor-dependent cell migration. J Biol Chem 287, 31321-9
[3] Fekete A, Bőgel G, Pesti S, Péterfi Z, Geiszt M, Buday L. (2013) EGF regulates tyrosine phosphorylation and membrane-translocation of the scaffold protein Tks5. J Mol Signal 8, 8
[4] Ádám C, Fekete A, Bőgel G, Németh Z, Tőkési N, Ovádi J, Liliom K, Pesti S, Geiszt M, Buday L. Accumulation of the PX domain mutant Frank-ter Haar syndrome protein Tks4 in aggresomes. (20115) Cell Commun Signal. 2015 13, 33
[5] Dülk M, Kudlik G, Fekete A, Ernszt D, Kvell K, Pongrácz JE, Merő BL, Szeder B, Radnai L, Geiszt M, Csécsy D, Kovács T, Uher F, Lányi Á, Vas V, Buday L. (2016) The scaffold protein Tks4 is required for the differentiation of mesenchymal stromal cells (MSCs) into adipogenic and osteogenic lineages. Sci Rep. 2016 6, 34280
[6] Vas V, Háhner T, Kudlik G, Fekete A, Ernszt D, Kvell K, Kuti D, Kovács K.J., Tóvári J, Trexler M, Merő BL, Szeder B, Koprivanacz K, Buday L. (2019) Analysis of Tks4 Knockout Mice Suggests a Role for Tks4 in Adipose Tissue Homeostasis in the Context of Beigeing. Cells. 2019 8, 831
[7] Vas V, Kovács T, Körmendi Sz, Bródy A, Kudlik Gy, Szeder B, Mező D, Kállai D, Koprivanacz K, Merő BL, Dülk M, Tóvári J, Vajdovich P, Neslihan SS, Özcan I, Helyes Zs, Dobó-Nagy Cs, Buday L. (2019) Significance of the Tks4 scaffold protein in bone tissue homeostasis. Sci Rep. 2019 9, 5781
[8] Dülk M, Szeder B, Glatz G, Merő BL, Koprivanacz K, Kudlik G, Vas V, Sipeki S, Cserkaszky A, Radnai L, Buday L.(2018) EGF Regulates the Interaction of Tks4 with Src through Its SH2 and SH3 Domains. Biochemistry. 2018 57(28), 4186-4196.
[9] G Kudlik, T Takács, L Radnai, A Kurilla, B Szeder, K Koprivanacz, Merő BL, Buday L, Vas V. (2020) Advances in understanding TKS4 and TKS5: molecular scaffolds regulating cellular processes from podosome and invadopodium formation to differentiation and tissue homeostasis. International Journal of Molecular Sciences 2020, 21 (21), 8117
- A Caskin állványfehérjék vizsgálata
A Caskin (CASK-interacting) állványfehérjék szerkezetében megtalálhatóak az ankyrin-ismétlődések, a SAM és SH3 domének, valamint számos prolinban-gazdag régió. A Caskin1 és a Caskin2 fehérje szerkezete elsősorban a prolinban-gazdag régió hosszában tér el. Ugyanakkor expressziójukban jelentősebb a különbség: míg a Caskin1 kizárólag a központi idegrendszerben expresszálódik, addig a Caskin2 több szövetben, illetve szervben is megjelenik. Tompa Péter csoportjával együttműködésben kimutatták, hogy a Caskin1 fehérje szerkezete jelentős részben rendezetlen. Ugyancsak kimutatták, hogy a prolinban-gazdag rendezetlen régió funkcióképes, ugyanis megköti SH3 doménjén keresztül az Abi2 (Abl-interactor 2) állványfehérjét [1]. Az EphB1 receptor tirozin kináz jelátviteli jelpályáját vizsgálva kimutatták, hogy az autofoszforilálódott receptor képes komplexet képezni a Caskin1-gyel, az Nck kapcsoló fehérjén keresztül. A receptorhoz kötődő Caskin1 ezt követően az SH3 doménjében található két tirozin oldalláncon foszforilálódik [2]. Az elmúlt években a csoport előállította a Caskin1/Caskin2 dupla génhiányos egértörzset a fehérje élettani szerepének további vizsgálatához [3].
[1] Balazs A, Csizmok V, Buday L, Rakacs M, Kiss R, Bokor M, Udupa R, Tompa K, Tompa P. (2009) High levels of structural disorder in scaffold proteins as exemplified by a novel neuronal protein, cask-interactive protein1. FEBS J 276, 4168
[2] Pesti S, Balázs A, Udupa R, Szabó B, Fekete A, Bögel G and Buday L. (2012) Complex formation of EphB1/NCK/Caskin1 leads to tyrosine phosphorylation and structural changes of the Caskin1 SH3 domain. Cell Commun Signal 10, 36
[3] N Bencsik, S Pusztai, S Borbély, A Fekete, M Dülk, V Kis, S Pesti, V Vas, Szűcs A, Buday L, Schlett K. (2019) Dendritic spine morphology and memory formation depend on postsynaptic Caskin proteins. Sci Rep. 2019 9, 16843
- SH3 domének tirozin foszforilációja, mint a fehérje-fehérje interakció egyik új szabályozási lehetősége
A Buday László vezette csoport az elmúlt 20 évben intenzíven vizsgálta a fehérjékben található SH3 domének működését. Így tanulmányoztak olyan SH3 domént tartalmazó fehérjéket, mint a Grb2, a Crk, az Nck, a Vav2, a cortactin, az Lck, az SLP-76, a Caskin1, az Src, vagy a Tks4 és Tks5 fehérjék [1-8]. Egyik jelentős felfedezésük, hogy bizonyos esetekben az SH3 domén tirozin foszforilácó tárgya lehet [9], ami befolyásolhatja az általa közvetített fehérje-fehérje interakciót. Jelenleg a csoport intenzíven vizsgálja az SH3 domének tirozin foszforilációjának biokémiai sajátságait [10,11].
[1] Buday, L., Khwaja, A., Sipeki, S., Faragó, A., and Downward, J. (1996) Interactions of Cbl with two adaptor proteins, Grb2 and Crk, upon T cell activation. J Biol Chem 271, 6159-6163
[2] Wunderlich, L., Faragó, A., and Buday, L. (1999) Characterization of interactions of Nck with Sos and dynamin. Cellular Signalling 11,25-29
[3] Wunderlich, L., Gohér, A., Faragó, A., Downward, J., and Buday, L. (1999) Requirement of multiple SH3 domains of Nck for ligand binding. Cellular Signalling 11, 253-262
[4] Buday, L. (1999) Membrane-targeting of signalling molecules by SH2/SH3 domain-containinmg adaptor proteins. Biochim Biophys Acta 1422,187-204
[5] Tamas P, Solti Z, Bauer P, Illes A, Sipeki S, Bauer A, Farago A, Downward J, Buday L. (2003) Mechanism of EGF regulation of Vav2, a guanine nucleotide exchange factor for Rac. J Biol Chem 278, 5163-5171
[6] Illes A, Enyedi B, Tamas P, Balazs A, Bogel G, Lukacs M, Buday L. (2006) Cortactin is required for integrin-mediated cell spreading. Immunol Letters 104, 124-130
[7] S Sipeki, K Koprivanacz, T Takács, A Kurilla, L László, V Vas, L Buday. (2021) Novel Roles of SH2 and SH3 Domains in Lipid Binding, Cells. 10 (5), 1191
[8] Hem CD, Sundvold-Gjerstad V, Granum S, Koll L, Abrahamsen G, Buday L, Spurkland A. (2015) T cell specific adaptor protein (TSAd) promotes interaction of Nck with Lck and SLP-76 in T cells. Cell Commun Signal 13, 31
[9] Pesti S, Balázs A, Udupa R, Szabó B, Fekete A, Bögel G and Buday L. (2012) Complex formation of EphB1/NCK/Caskin1 leads to tyrosine phosphorylation and structural changes of the Caskin1 SH3 domain. Cell Commun Signal 10, 36
[10] B Merő, L Radnai, G Gógl, O Tőke, I Leveles, K Koprivanacz, B Szeder, Dülk M, Kudlik G, Vas V, Cserkaszky A, Sipeki Sz, Nyitray L, Vértessy BG, Buday L. (2019) Structural insights into the tyrosine phosphorylation–mediated inhibition of SH3 domain–ligand interactions. Journal of Biological Chemistry 294 (12), 4608-4620
[11] B Merő, K Koprivanacz, A Cserkaszky, L Radnai, V Vas, G Kudlik, G Gógl, Sok P, Póti LÁ, Szeder B, Nyitray L, Reményi A, Geiszt M, Buday L. Characterization of the Intramolecular Interactions and Regulatory Mechanisms of the Scaffold Protein Tks4. International journal of molecular sciences 22 (15), 8103
- Genom annotáció
A 21. században az orvostudomány, gyógyszerfejlesztés, mezőgazdaság, biotechnológia valamint az evolúcióbiológiai tudományok egyre inkább a genom projektekből származó adatokra támaszkodnak. A genom-szekvencia értelmezésének egyik legdöntőbb lépése a fehérjekódoló gének azonosítása és szerkezetük meghatározása bioinformatikai eszközökkel. A genom szekvenciákat hasznosító biológiai kutatások legtöbb további lépésének sikere ezeknek az adatoknak a minőségétől függ. A génazonosítás nehézségeit illusztrálhatjuk azzal, hogy – a génpredikciós módszerek jelentős fejlődése ellenére – a fehérjekódoló gének szerkezetének predikciója nem kellően megbízható: a becslések szerint a megjósolt gének kevesebb, mint 50%-ának helyes a szerkezete [1].
Ennek a problémának a megoldása érdekében egy olyan módszert dolgoztunk ki, amely lehetővé teszi annak eldöntését, hogy egy kísérletesen meghatározott vagy in silico jósolt fehérjekódoló szekvencia hibás-e (abnormális, fragmentum, tévesen megjósolt). A MisPred megközelítés alapja az az általános megfontolás, hogy egy fehérjekódoló gén megjósolt szerkezete valószínűleg téves, ha a gén (vagy az általa kódolt fehérje) valamely tulajdonsága nem egyeztethető össze a fehérjekódoló génekre és a fehérjékre jellemző törvényszerűségek valamelyikével [2-4].
[3] Nagy A, Hegyi H, Farkas K, Tordai H, Kozma E, Bányai L and Patthy L (2008) Quality control of gene predictions. In: Frishman D., Valencia A. (eds) Modern Genome Annotation. Springer, Vienna.
A hibás gének/fehérjék azonosítása azért lényeges, mert ezzel megakadályozhatjuk, hogy a téves adatokból téves következtetést vonjanak le a kutatók [5-7].
Mivel a szekvencia adatbázisok jelentős mértékben „szennyezettek” hibás szekvenciákkal, a hibás szekvenciák azonosítása és kijavítása csak automatizált minőségellenőrző és hibajavító módszerekkel történhet. A FixPred projekt keretében hatékony eszközöket fejlesztettünk ki a hibás szekvenciák automatizált kijavítására [8-9].
A genom annotáció következő lépése a különböző gének/fehérjék molekuláris funkcióinak, szerkezetének és biológiai szerepeinek jóslása/meghatározása. Mindez nélkülözhetetlen a gének orvostudományi, mezőgazdasági vagy biotechnológiai jelentőségének megértéséhez. A genom annotáció során azonosított fehérjék szerkezetének, funkciójának és biológiai szerepének jóslását komplex bioinformatikai elemzések segítségével végezzük.
Ennek során az új fehérjéket/géneket az ismert doméncsaládokba soroljuk be, megjósoljuk legvalószínűbb biológiai funkciójukat, sejten belüli lokalizációjukat és szerkezet-funkció összefüggéseiket. Ilyen vizsgálatokkal jellemeztünk több orvosbiológiai szempontból fontos multidomén fehérjét (pl. WFIKKN1, WFIKKN2 fehérjék [10-11] és több új fehérje doméncsaládot (pl. NTR-domének [12], PAN-domének [13], WIF-domének [14], LCCL-domének [15].
[14] Patthy L (2000) The WIF module. Trends Biochem Sci 25(1):12-13.
[15] Trexler M, Bányai L and Patthy L (2000) The LCCL module. Eur J Biochem 267(18):5751-5757.
Gottfried Otting NMR csoportjával (Research School of Chemistry, The Australian National University, Canberra, Australia) együttműködésben, elsőként határoztuk meg egy LCCL-domén [16], egy NTR-domén [17] és egy WIF-domén [18] szerkezetét, igazolva az in silico szerkezet-predikciók helyességét.
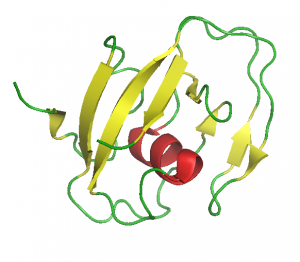
LCCL-domén (1JBI.pdb)
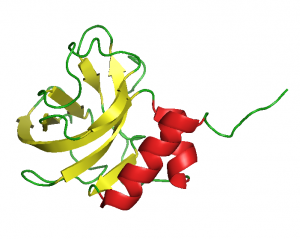
NTR-domén (1UAP.pdb)
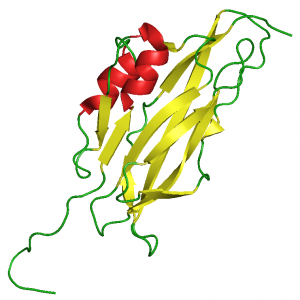
WIF-domén (2D3J.pdb)
Különböző élőlények genom, transzkriptóm és proteóm adatainak elemzésével új bizonyítékokat szolgáltattunk arra, hogy az exon-shuffling és modul-shuffling kulcszerepet játszott a metazoa-típusú multicellularitáshoz nélkülözhetetlen fehérje eszköztár kialakulásában [19-21].
Humán tumorok genomadatainak elemzése alapján új eljárást dolgoztunk ki azoknak a géneknek az azonosítására, melyek kulcsszerepet játszanak a tumorok kialakulásában és evolúciójában [22].
- Orvosbiológiai szempontból fontos fehérjék szerkezetének és funkciójának kísérletes vizsgálata
In silico fehérje/domén jóslásaink eredményét kísérletes technikákkal kapcsoltuk össze, hogy meghatározzuk néhány orvostudományi szempontból fontos multidomén fehérje szerkezetét, valamint molekuláris és biológiai funkcióját. Ennek a megközelítésnek egyik példája az izomfejlődés szabályozásában szerepet játszó WFIKKN1 és WFIKKN2 fehérje, melyek felfedezése és jellemzése elsősorban csoportunk munkájának köszönhető [1-2].
Kimutattuk, hogy mindkét WFIKKN fehérje hatékony inhibitora a közeli rokon GDF8 (miosztatin) és GDF11 növekedési faktoroknak, de nem gátolják a TGFβ család más tagjainak szignál transzdukciós aktivitását [3-7].
Eredményeink jelentőségét az adja meg, hogy a WFIKKN1 és WFIKKN2 fehérjék és/vagy származékaik – mint miosztatin-antagonisták – alkalmasak lehetnek az izomvesztéssel járó betegségek kezelésére. A Fehérjetudomány és alkalmazásai Nemzeti Program keretében olyan molekulák kifejlesztésén dolgozunk, melyek erősebben és specifikusabban kötődnek a miosztatinhoz, így hatékonyabban gátolják a miosztatin aktivitását.
Hasonlóképpen fontos orvosbiológiai szempontból a Wnt Inhibitory Factor 1 fehérje, minthogy a WIF1 gén epigenetikai elnémítása (és ezáltal a Wnt- β catenin útvonal rendellenes aktiválása) gyakran áll a daganatkialakulás hátterében. Rekombináns WIF1 fehérje alkalmazásával gátolni lehet a tumor progressziót, azonban a célzott terápiánál figyelembe kell venni azt a tényt, hogy eltérő mértékben képes a különböző Wnt-k gátlására [8-9] továbbá, hogy a WIF1 fehérje a hedgehog-útvonal szabályozásában is szerepet játszik [10].
A Fehérjetudomány és alkalmazásai Nemzeti Program keretében – a WIF-domén szerkezetének ismeretében – olyan WIF1 mutánsok előállításán dolgozunk, melyek a különböző Wnt-khez nagyobb affinitással és specifikusabban kötődnek és így hatékonyabban alkalmazhatók tumorspecifikus terápiás eljárásokban.
Főbb műszerek, berendezések, szolgáltatások
Sejt-tenyésztő laboratórium
Excella ECO-170 CO2 inkubátor
ESCO CLASS II BSC inkubátor
NÜVE ES110 inkubátor
Zeiss AXIO fáziskontraszt mikroszkóp
Applied Biosystem Quantstudio 6 pro RT-qPCR instrument
Fehérjék tisztítása, jellemzése:
CARY 300 UV-VIS spektrofotométer
CARY Eclipse spektrofluotiméter
UNICORN FPLC készülék
ChemiImager 5500 géldokumentációs rendszer
LSM710 konfokális mikroszkóp
Oktatási tevékenység
Buday László részállású egyetemi tanárként oktat a Semmelweis Egyetem Általános Orvosi Karán Orvosi Biokémiát. MSc képzés keretében kurzust tart a Pázmány Péter Katolikus Egyetem Információs Technológiai Karán Orvosi biotechnológia tárgyból, továbbá kurzust tart az ELTE Biológiai Doktori Iskola Szerkezeti Biológia programjában.
Csoportvezető
Buday László