Kutatócsoport-vezető
Orbán Tamás, Ph.D.
Bemutatkozás, általános kutatási terület
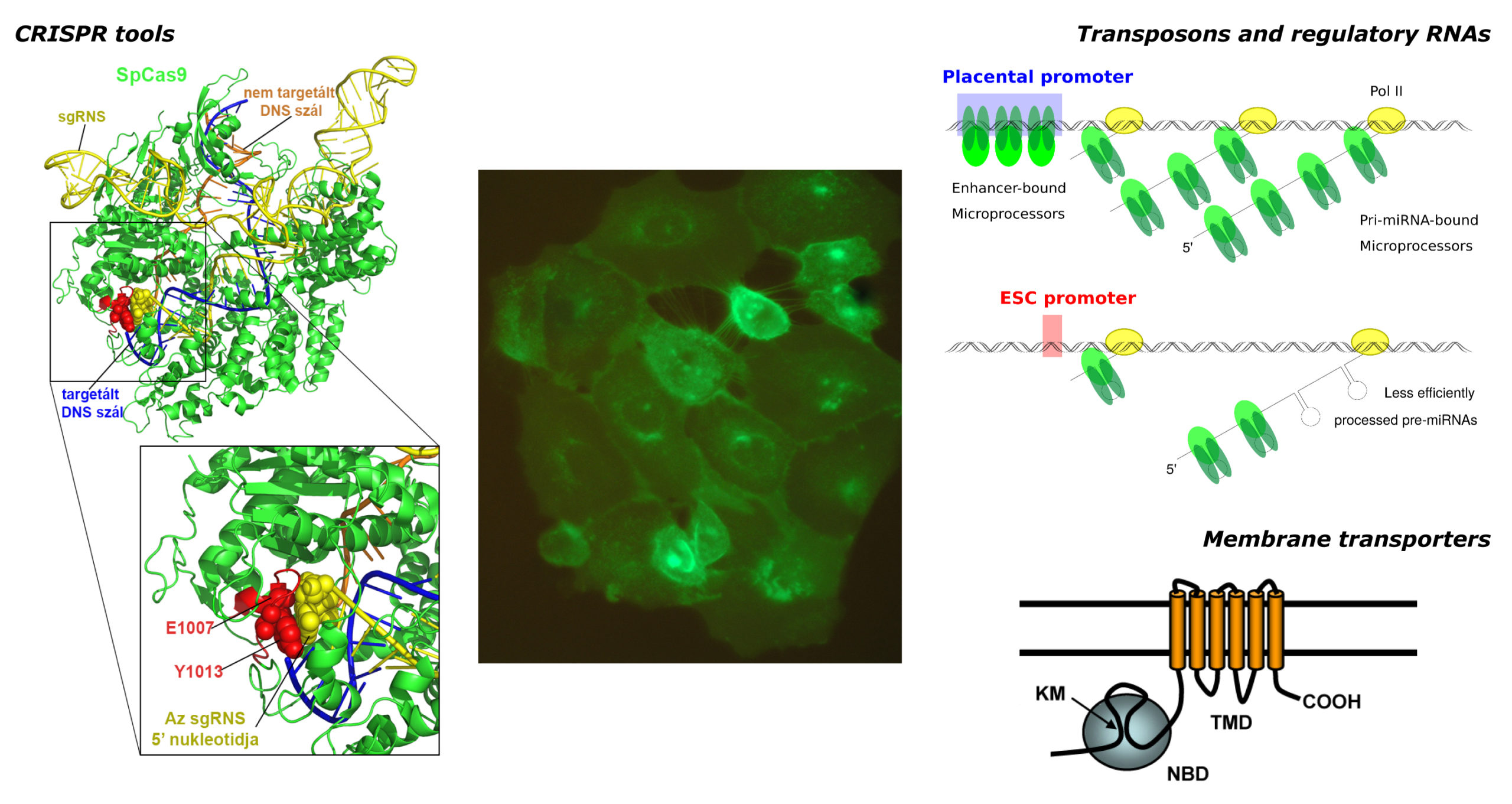
A Génreguláció Kutatócsoporton belül három, egymással szorosan együttműködő kutatólaboratórium működik, a Transzpozonok és szabályozó RNS-ek Laboratórium (vezetője Orbán Tamás), a CRISPR Laboratórium (vezetője Welker Ervin) és a Membrán Transzporter Laboratórium (vezetője Sarkadi Balázs és Telbisz Ágnes). A korábbi Biomembrán Csoport kutatási hagyományaira építve a csoportok jelenleg a génexpresszió szabályozásának különböző aspektusait kutatják, illetve különféle molekuláris biológiai esszéket és vizsgálati módszereket fejlesztenek, amelyek humán betegségmodellek felállításában fontos szerepet játszanak.
A Molekuláris Élettudományi Intézetben több együttműködő csoport közül kiemelt partnerünk a Molekuláris Sejtbiológia Kutatócsoport (vezetője Homolya László), akikkel különösen a humán őssejtbiológia területén (laboratóriumvezető Apáti Ágota) sok közös projektben veszünk részt. Mindemellett igyekszünk kihasználni az HUN-REN – TTK által nyújtotta egyedülálló lehetőségeket, hogy együtt dolgozzunk különböző kutatócsoportokkal a kémia, a biológia és a kísérleti pszichológia területéről.
Transzpozonok és szabályozó RNS-ek Laboratórium
Vezetője: Dr. Orbán Tamás – tudományos főmunkatárs (publikációk)
A laboratórium egyik fő kutatási területe az eukarióta DNS transzpozonok működésének vizsgálata emlős rendszerekben. Az endogén szabályozási folyamatok megértése mellett kíváncsiak vagyunk arra, hogy milyen molekuláris szintű védekezési folyamatok léteznek DNS transzpozonok ellen, és hogyan történhet a DNS transzpozonok domesztikációja, elsősorban az emberi genomban. Kutatásaink egyik fontos iránya továbbá a transzpozon-alapú génbeviteli eljárások fejlesztése: legfőképpen a Sleeping Beauty és a piggyBac transzpozonos rendszerek alkalmazása és hatékonyságának analízise emlős modellrendszerekben, beleértve az embrionális őssejteken történő alkalmazások vizsgálatát is.
A laboratórium másik fontos kutatási területe az RNS interferencia folyamatainak tanulmányozása, ezen belül is a mikroRNS molekulák érési folyamatainak vizsgálata. A molekuláris folyamatok részleteinek feltárása kapcsán kutatásaink középpontjában egyrészt a mikroRNS klaszterek regulációjának, másrészt az emlős mirtron útvonal szabályozási részleteinek a feltárása áll. Az utóbbi alternatív érési útvonal felhasználásával mindemellett olyan mesterséges mirtron eredetű mikroRNS molekulákat igyekszünk előállítani, amelyek később akár terápiás célokra is alkalmazhatóak lennének.
Csoportkép (2018): Csoportkép (2022):


Végzett PhD hallgatók:
- Schamberger Anita (2014)
- Kolacsek Orsolya (2017)
- Sándor Sára Anna (2018)
- Fóthi Ábel (2023)
- Wachtl Gerda Gabriella (2023)
Fontosabb publikációk:
Orbán TI: One locus, several functional RNAs-emerging roles of the mechanisms responsible for the sequence variability of microRNAs. Biologia Futura. 2023, 74(1-2):17-28.
Gyöngy Z, Mocsár G, Hegedűs É, Stockner T, Ritter Z, Homolya L, Schamberger A, Orbán TI, Remenyik J, Szakacs G, Goda K: Nucleotide binding is the critical regulator of ABCG2 conformational transitions. eLife. 2023, 12:e83976.
Reé D, Fóthi Á, Varga N, Kolacsek O, Orbán TI, Apáti Á: Partial disturbance of Microprocessor function in human stem cells carrying a heterozygous mutation in the DGCR8 gene. Genes. 2022, 13(11):1925. (cover page story)
Raskó T, Pande A, Radscheit K, Zink A, Singh M, Sommer C, Wachtl G, Kolacsek O, Inak G, Szvetnik A, Petrakis S, Bunse M, Bansal V, Selbach M, Orbán TI, Prigione A, Hurst LD, Izsvák Z: A novel gene controls a new structure: PiggyBac Transposable Element-Derived 1, unique to mammals, controls mammal-specific neuronal paraspeckles. Molecular Biology and Evolution. 2022, 39(10):msac175.
Wachtl G, Schád É, Huszár K, Palazzo A, Ivics Z, Tantos Á, Orbán TI: Functional characterization of the N-terminal disordered region of the piggyBac transposase. International Journal of Molecular Sciences. 2022, 23(18):10317.
Kolacsek O, Wachtl G, Fóthi Á, Schamberger A, Sándor S, Pergel E, Varga N, Raskó T, Izsvák Z, Apáti Á, Orbán TI: Functional indications for transposase domestications – characterization of the human piggyBac transposase derived (PGBD) activities. Gene. 2022, 834:146609.
Fóthi Á, Biró O, Erdei Z, Apáti Á, Orbán TI: Tissue-specific and transcription-dependent mechanisms regulate primary microRNA processing efficiency of the human chromosome 19 MicroRNA cluster. RNA Biology. 2021, 18(8):1170-1180.
Schamberger A, Várady G, Fóthi Á, Orbán TI: Posttranscriptional Regulation of the Human ABCG2 Multidrug Transporter Protein by Artificial Mirtrons. Genes. 2021, 12(7): 1068.
Reé D, Borsy A, Fóthi Á, Orbán TI, Várady G, Erdei Z, Sarkadi B, Réthelyi J, Varga N, Apáti Á: Establishing a human embryonic stem cell clone with a heterozygous mutation in the DGCR8 gene. Stem Cell Research. 2020, 50:102134.
Zámbó B, Mózner O, Bartos Z, Török G, Várady G, Telbisz Á, Homolya L, Orbán TI, Sarkadi B: Cellular expression and function of naturally occurring variants of the human ABCG2 multidrug transporter. Cellular and Molecular Life Sciences. 2020, 77(2):365-378.
Biró O, Fóthi Á, Alasztics B, Nagy B, Orbán TI, Rigó J Jr.: Circulating exosomal and Argonaute-bound microRNAs in preeclampsia. Gene. 2019, 692:138-144.
Kolacsek O, Orbán TI: Transcription activity of transposon sequence limits Sleeping Beauty transposition. Gene. 2018, 676:184-188.
Szádeczky-Kardoss I, Csorba T, Auber A, Schamberger A, Nyikó T, Taller J, Orbán TI, Burgyán J, Silhavy D: The nonstop decay and the RNA silencing systems operate cooperatively in plants. Nucleic Acids Research. 2018, 46(9):4632-4648.
Kolacsek O, Pergel E, Varga N, Apáti Á, Orbán TI: Ct shift: A novel and accurate real-time PCR quantification model for direct comparison of different nucleic acid sequences and its application for transposon quantifications. Gene. 2017, 598:43-49.
Sándor S, Jordanidisz T, Schamberger A, Várady G, Erdei Z, Apáti Á, Sarkadi B, Orbán TI: Functional characterization of the ABCG2 5′ non-coding exon variants: Stem cell specificity, translation efficiency and the influence of drug selection. Biochimica et Biophysica Acta – Gene Regulatory Mechanisms. 2016, 1859(7):943-51.
Szebényi K, Füredi A, Kolacsek O, Pergel E, Bősze Z, Bender B, Vajdovich P, Tóvári J, Homolya L, Szakács G, Héja L, Enyedi Á, Sarkadi B, Apáti Á, Orbán TI: Generation of a Homozygous Transgenic Rat Strain Stably Expressing a Calcium Sensor Protein for Direct Examination of Calcium Signaling. Scientific Reports. 2015, 5:12645.
Szebényi K, Füredi A, Kolacsek O, Csohány R, Prókai Á, Kis-Petik K, Szabó A, Bősze Z, Bender B, Tóvári J, Enyedi Á, Orbán TI, Apáti Á, Sarkadi B: Visualization of calcium dynamics in rat kidney proximal tubules. Journal of the American Society of Nephrology. 2015, 26(11):2731-40.
Schamberger A, Orbán TI: 3’ isomiR species and DNA contamination influence reliable quantification of microRNAs by stem-loop quantitative PCR. PLoS ONE. 2014, 9(8): e106315.
Kolacsek O, Erdei Z, Apáti A, Sándor S, Izsvák Z, Ivics Z, Sarkadi B, Orbán TI: Excision efficiency is not strongly coupled to transgenic rate: cell type dependent transposition efficiency of Sleeping Beauty and piggyBac DNA transposons. Human Gene Therapy Methods. 2014, 25(4):241-52.
Anita Schamberger and Tamás I. Orbán: Experimental validation of predicted mammalian miRNAs of mirtron origin. In: RNA Mapping, Methods and Protocols, Methods in Molecular Biology, Lucrecia Alvarez and Mahtab Nourbakhsh (Eds.). Springer Verlag, ISBN: 978-1-4939-1061-8, 2014; 1182:245-63.
Orsolya Kolacsek, Zsuzsanna Izsvák, Zoltán Ivics, Balázs Sarkadi, Tamás I. Orbán: Quantitative analysis of DNA transposon-mediated gene delivery: the Sleeping Beauty system as an example. In: Genomics III – Methods, Techniques and Applications, iConcept Press Ltd Book, ISBN: 978-1-922227-096, 2014, 97-123.
Schamberger A, Sarkadi B, Orban TI: Human mirtrons can express functional microRNAs simultaneously from both arms in a flanking exon-independent manner. RNA Biology. 2012, 9(9):1177-85. (cover page story)
Kolacsek O, Krízsik V, Schamberger A, Erdei Z, Apáti Á, Várady G, Mátés L, Izsvák Z, Ivics Z, Sarkadi B, Orbán TI: Reliable transgene-independent method for determining Sleeping Beauty transposon copy numbers. Mobile DNA. 2011, 2(1):5.
Tamás I. Orbán, Ágota Apáti, Zsuzsanna Izsvák, Zoltán Ivics and Balázs Sarkadi: Use of Transposon-Transposase Systems for Stable Genetic Modification of Embryonic Stem Cells. In: Methodological Advances in the Culture, Manipulation and Utilization of Embryonic Stem Cells for Basic and Practical Applications, Craig Atwood (Ed.). InTech, ISBN: 978-953-307-197-8; 2011: 259-274
Sarkadi B, Orbán TI, Szakács G, Várady G, Schamberger A, Erdei Z, Szebényi K, Homolya L, Apáti A: Evaluation of ABCG2 expression in human embryonic stem cells: crossing the same river twice? Stem Cells. 2010, 28(1):174-6.
Orbán TI, Apáti A, Németh A, Varga N, Krizsik V, Schamberger A, Szebényi K, Erdei Z, Várady G, Karászi E, Homolya L, Német K, Gócza E, Miskey C, Mátés L, Ivics Z, Izsvák Z, Sarkadi B: Applying a “double-feature” promoter to identify cardiomyocytes differentiated from human embryonic stem cells following transposon-based gene delivery. Stem Cells. 2009, 27(5):1077-87.
CRISPR Laboratórium
Vezető: Dr. Welker Ervin – tudományos tanácsadó (publikációk)
A laboratórium a CRISPR nukleázok működési mechanizmusát vizsgálja, genomszerkesztő eljárások hatékonyságának és pontosságának növelésén dolgozik. Kutatásuk elsősorban a Cas9 és Cas12a fehérjék működési módjának megértését tűzte ki célul, különös tekintettel a base editorokban és prime editorokban betöltött szerepükre. A csoport továbbá preklinikai eljárások kifejlesztésével foglalkozik genetikai betegségek génsebészeti gyógyítására összpontosítva.

Végzett PhD hallgatók:
- Tóth Eszter (2015)
- Nyeste Antal (2016)
- Cingaram Pradeep Kumar Reddy (2016)
- Kulcsár Péter István (2019)
- Weinhardt Nóra (2021)
- Tálas András (2021)
- Sudheer Babu Sangeetham (2022)
Fontosabb publikációk:
Péter, István Kulcsár; András, Tálas; Zoltán, Ligeti; Tóth, Eszter; Rakvács, Zsófia; Bartos, Zsuzsa; Sarah, Laura Krausz; Welker, Ágnes; Végi, Vanessza Laura, Huszár, Krisztina; Welker, Ervin A cleavage rule for selection of increased-fidelity SpCas9 variants with high efficiency and no detectable off-targets NATURE COMMUNICATIONS 14, 5746 (2023)
Huszár, Krisztina; Welker, Zsombor; Györgypál, Zoltán; Tóth, Eszter; Ligeti, Zoltán; Kulcsár, Péter István; Dancsó, János; Tálas, András; Krausz, Sarah Laura; Varga, Éva; Welker, Ervin Position-dependent sequence motif preferences of SpCas9 are largely determined by scaffold-complementary spacer motifs. NUCLEIC ACIDS RESEARCH gkad323 (2023)
Péter, István Kulcsár ; András, Tálas ; Zoltán, Ligeti ; Sarah, Laura Krausz ; Ervin, Welker SuperFi-Cas9 exhibits remarkable fidelity but severely reduced activity yet works effectively with ABE8e.
NATURE COMMUNICATIONS 13, 6858 (2022)
Simon, Dorottya Anna ; Tálas, András* ; Kulcsár, Péter István ; Biczók, Zsuzsanna ; Krausz, Sarah Laura ; Várady, György ; Welker, Ervin PEAR, a flexible fluorescent reporter for the identification and enrichment of successfully prime edited cells
ELIFE 11 Paper: e69504 , 19 p. (2022)
András Tálas, Dorottya A. Simon, Péter I. Kulcsár, Éva Varga, Sarah L. Krausz & Ervin Welker BEAR reveals that increased fidelity variants can successfully reduce the mismatch tolerance of adenine but not cytosine base editors NATURE COMMUNICATIONS volume 12, Article number: 6353 (2021)
Talas, Andras; Huszar, Krisztina; Péter István Kulcsár, Julia K Varga, Éva Varga, Eszter Tóth, Zsombor Welker, Gergely Erdős, Péter Ferenc Pach, Ágnes Welker, Zoltán Györgypál, Gábor E Tusnády, Ervin Welker A method for characterizing Cas9 variants via a one-million target sequence library of self-targeting sgRNAs NUCLEIC ACIDS RESEARCH gkaa1220 (2021)
Tóth, Eszter ; Varga, Éva ; Kulcsár, Péter István ; Kocsis-Jutka, Virág ; Krausz, Sarah Laura ; Nyeste, Antal ; Welker, Zsombor ; Huszár, Krisztina ; Ligeti, Zoltán ; Tálas, András ; Welker, Ervin Improved LbCas12a variants with altered PAM specificities further broaden the genome targeting range of Cas12a nucleases NUCLEIC ACIDS RESEARCH 2020 Paper: gkaa110 (2020)
Kulcsár, Péter István ; Tálas, András ; Tóth, Eszter ; Nyeste, Antal ; Ligeti, Zoltán ; Welker, Zsombor ; Welker, Ervin Blackjack mutations improve the on-target activities of increased fidelity variants of SpCas9 with 5′G-extended sgRNAs NATURE COMMUNICATIONS 11 : 1 Paper: 1223 (2020)
Tóth, Eszter ; Czene, Bernadett C ; Kulcsár, Péter I ; Krausz, Sarah L ; Tálas, András ; Nyeste, Antal ; Varga, Éva ; Huszár, Krisztina ; Weinhardt, Nóra ; Ligeti, Zoltán ; Adrienn É Borsy, Elfrieda Fodor, Ervin Welker Mb- and FnCpf1 nucleases are active in mammalian cells: activities and PAM preferences of four wild-type Cpf1 nucleases and of their altered PAM specificity variants. NUCLEIC ACIDS RESEARCH 46 : 19 pp. 10272-10285. , 14 p. (2018)
Tálas, András ; Péter, Istvan Kulcsár ; Nóra, Weinhardt ; Adrienn, Borsy ; Eszter, Tóth ; Kornélia, Szebényi ; Sarah, Laura Krausz ; Krisztina, Huszár ; István, Vida ; Ádám, Bianka Gordos, Orsolya Ivett Hoffman, Petra bencsura, Antal Nyeste, Zoltán Ligeti, Elfrieda Fodor, Ervin Welker A convenientmethod to pre-screen candidate guide RNAs for CRISPR/Cas9 gene editing by NHEJ-mediated integration of a ‘self-cleaving’ GFP-expression plasmid. DNA RESEARCH 24 : 6 pp. 609-621. , 13 p. (2017)
Kulcsar, PI ; Talas, A ; Huszar, K ; Ligeti, Z ; Toth, E ; Weinhardt, N ; Fodor, E ; Welker, E. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. GENOME BIOLOGY 18 Paper: 190, 17 p. (2017)
Toth, E ; Weinhardt, N ; Bencsura, P ; Huszar, K ; Kulcsar, PI ; Talas, A ; Fodor, E ; Welker, E. Cpf1 nucleases demonstrate robust activity to induce DNA modification by exploiting homology directed repair pathways in mammalian cells BIOLOGY DIRECT 11 Paper: 46 , 14 p. (2016)
Toth, E ; Huszar, K ; Bencsura, P ; Kulcsar, PI ; Vodicska, B ; Nyeste, A ; Welker, Z ; Toth, S ; Welker, E Restriction enzyme body doubles and PCR cloning: on the general use of type IIs restriction enzymes for cloning. PLOS ONE 9 : 3 Paper: e90896 , 12 p. (2014)
Membrán Transzporter Laboratórium
Vezetője: Prof. Dr. Sarkadi Balázs – emeritus professzor (publikációk)
A laboratórium elsősorban a membrántranszport fehérjék szerkezet-funkció összefüggéseit, a gyógyszerek hatásában és eloszlásában játszott szerepét vizsgálja. Egyik fontos kutatási területünk az „ATP Binding Cassette” (ABC) transzporterek vizsgálata, amelyek számos alapvető funkcióval rendelkeznek az emberi szervezetben. A multidrog rezisztens (MDR) ABC transzporterek komolyan veszélyeztetik a daganatellenes kemoterápia hatékonyságát, mindemellett kulcsfontosságú résztvevői a sejtszintű védekezésnek és a szöveti határfelületeken történő transzportnak.
Az ABC transzporterek szerepe a gyógyszerek felszívódása, eloszlása, anyagcseréje, kiválasztása és toxicitása (ADME-Tox) tekintetében széles körben bizonyított. A jelenlegi gyógyszerfejlesztés is megköveteli a potenciális új hatóanyagok kölcsönhatásainak részletes vizsgálatát a gyógyszer-transzporterekkel. Laboratóriumunk jelenleg néhány kulcsfontosságú ABC transzporter expressziójára, jellemzésére, izolálására és funkcionális jellemzésére fókuszál, beleértve az ABCG2 (BCRP, MXR), ABCB1 (Pgp, MDR1), ABCB11 (epesót szállító fehérje, BSEP), és az ABCB6 fehérjét.
A laboratórium tagjai ugyancsak aktívan vizsgálják az emberi sejtek kalcium transzporter fehérjéit, azok esetleges szerepét a betegségekben. Funkcionális teszt-rendszereket hozunk létre és nagy kapacitású farmakológiai és toxikológiai esszéket fejlesztünk ki. Az utóbbi években széleskörű együttműködésekben vizsgáljuk a membrántranszporterek expresszióját, genetikai polimorfizmusait, szerepüket az emberi őssejtek differenciációjában és védekezésében. A COVID-19 pandémia hatására fontos új kutatási projektünk a SARS-CoV-2 vírus fehérjéinek és a vírus ellen kifejlesztett gyógyszereknek a transzporter fehérjékkel lehetséges kölcsönhatásainak vizsgálata.
Végzett PhD hallgatók:
- Hegedüs Csilla (2014)
- Szebényi Kornélia (2015)
- Antalffy Géza (2015)
- Erdei Zsuzsa (2016)
- Varga Nóra (2017)
- Zámbó Boglárka (2018)
- Temesszentandrási-Ambrus Csilla (2023)
Fontosabb publikációk:
Mózner O, Moldvay J, Szabó KS, Vaskó D, Domján J, Ács D, Ligeti Z, Fehér C, Hirsch E, Puskás L, Stahl C, Frey M, Sarkadi B. Application of a Receptor-Binding-Domain-Based Simple Immunoassay for Assessing Humoral Immunity against Emerging SARS-CoV-2 Virus Variants. Biomedicines. 2023 Dec 1;11(12):3193. doi: 10.3390/biomedicines11123193.
Broca-Brisson L, Harati R, Disdier C, Mozner O, Gaston-Breton R, Maïza A, Costa N, Guyot AC, Sarkadi B, Apati A, Skelton MR, Madrange L, Yates F, Armengaud J, Hamoudi R, Mabondzo A. Deciphering neuronal deficit and protein profile changes in human brain organoids from patients with creatine transporter deficiency. Elife. 2023 Oct 13;12:RP88459. doi: 10.7554/eLife.88459.
Mózner O, Zámbó B, Bartos Z, Gergely A, Szabó KS, Jezsó B, Telbisz Á, Várady G, Homolya L, Hegedűs T, Sarkadi B. Expression, Function and Trafficking of the Human ABCG2 Multidrug Transporter Containing Mutations in an Unstructured Cytoplasmic Loop. Membranes (Basel). 2023 Oct 4;13(10):822. doi: 10.3390/membranes13100822.
Pálinkás M, Szabó E, Kulin A, Mózner O, Rásonyi R, Juhász P, Nagy K, Várady G, Vörös D, Zámbó B, Sarkadi B, Poór G. Genetic polymorphisms and decreased protein expression of ABCG2 urate transporters are associated with susceptibility to gout, disease severity and renal-overload hyperuricemia. Clin Exp Med. 2023 Aug;23(4):1277-1284. doi: 10.1007/s10238-022-00848-7.
Bakos É, Temesszentandrási-Ambrus C, Özvegy-Laczka C, Gáborik Z, Sarkadi B, Telbisz Á. Interactions of the Anti-SARS-CoV-2 Agents Molnupiravir and Nirmatrelvir/Paxlovid with Human Drug Transporters. Int J Mol Sci. 2023 Jul 8;24(14):11237. doi: 10.3390/ijms241411237.
Kulin A, Kucsma N, Bohár B, Literáti-Nagy B, Korányi L, Cserepes J, Somogyi A, Sarkadi B, Szabó E, Várady G. Genetic Modulation of the GLUT1 Transporter Expression-Potential Relevance in Complex Diseases. Biology (Basel). 2022 Nov 16;11(11):1669. doi: 10.3390/biology11111669.
Szabó E, Kulin A, Jezsó B, Kucsma N, Sarkadi B, Várady G. Selective Fluorescent Probes for High-Throughput Functional Diagnostics of the Human Multidrug Transporter P-Glycoprotein (ABCB1). Int J Mol Sci. 2022 Sep 13;23(18):10599. doi: 10.3390/ijms231810599.
Mioč M, Telbisz Á, Radman K, Bertoša B, Šumanovac T, Sarkadi B, Kralj M. Interaction of crown ethers with the ABCG2 transporter and their implication for multidrug resistance reversal. Histochem Cell Biol. 2022 Sep;158(3):261-277. doi: 10.1007/s00418-022-02106-z. Epub 2022 Jun 1.
Ferenci T, Sarkadi B. RBD-specific antibody responses after two doses of BBIBP-CorV (Sinopharm, Beijing CNBG) vaccine. BMC Infect Dis. 2022 Jan 24;22(1):87. doi: 10.1186/s12879-022-07069-z.
Szabó E, Kulin A, Mózner O, Korányi L, Literáti-Nagy B, Vitai M, Cserepes J, Sarkadi B, Várady G. Potential role of the ABCG2-Q141K polymorphism in type 2 diabetes. PLoS One. 2021 Dec 2;16(12):e0260957. doi: 10.1371/journal.pone.0260957. eCollection 2021.
Ambrus C, Bakos É, Sarkadi B, Özvegy-Laczka C, Telbisz Á. Interactions of anti-COVID-19 drug candidates with hepatic transporters may cause liver toxicity and affect pharmacokinetics. Sci Rep. 2021 Sep 8;11(1):17810. doi: 10.1038/s41598-021-97160-3. PMID: 34497279; PMCID: PMC8426393.
Mózner O, Zámbó B, Sarkadi B. Modulation of the Human Erythroid Plasma Membrane Calcium Pump (PMCA4b) Expression by Polymorphic Genetic Variants. Membranes (Basel). 2021 Jul 30;11(8):586. doi: 10.3390/membranes11080586. PMID: 34436349; PMCID: PMC8401972.
Nagy T, Tóth Á, Telbisz Á, Sarkadi B, Tordai H, Tordai A, Hegedűs T. The transport pathway in the ABCG2 protein and its regulation revealed by molecular dynamics simulations. Cell Mol Life Sci. 2021 Mar;78(5):2329-2339. doi: 10.1007/s00018-020-03651-3. Epub 2020 Sep 26. PMID: 32979053; PMCID: PMC7966132.
Szabó E, Kulin A, Korányi L, Literáti-Nagy B, Cserepes J, Somogyi A, Sarkadi B, Várady G. Alterations in erythrocyte membrane transporter expression levels in type 2 diabetic patients. Sci Rep. 2021 Feb 2;11(1):2765. doi: 10.1038/s41598-021-82417-8. PMID: 33531564; PMCID: PMC7854743.
Telbisz Á, Ambrus C, Mózner O, Szabó E, Várady G, Bakos É, Sarkadi B, Özvegy- Laczka C. Interactions of Potential Anti-COVID-19 Compounds with Multispecific ABC and OATP Drug Transporters. Pharmaceutics. 2021 Jan 9;13(1):81. doi: 10.3390/pharmaceutics13010081. PMID: 33435273; PMCID: PMC7827085.
Sarkadi B, Homolya L, Hegedűs T. The ABCG2/BCRP transporter and its variants – from structure to pathology. FEBS Lett. 2020 Dec;594(23):4012-4034. doi: 10.1002/1873-3468.13947. Epub 2020 Oct 16. PMID: 33015850.
Kovacsics D, Brózik A, Tihanyi B, Matula Z, Borsy A, Mészáros N, Szabó E, Németh E, Fóthi Á, Zámbó B, Szüts D, Várady G, Orbán TI, Apáti Á, Sarkadi B. Precision-engineered reporter cell lines reveal ABCG2 regulation in live lung cancer cells. Biochem Pharmacol. 2020 May;175:113865. doi: 10.1016/j.bcp.2020.113865. Epub 2020 Mar 4. PMID: 32142727.
Zámbó B, Mózner O, Bartos Z, Török G, Várady G, Telbisz Á, Homolya L, Orbán TI, Sarkadi B. Cellular expression and function of naturally occurring variants of the human ABCG2 multidrug transporter. Cell Mol Life Sci. 2020 Jan;77(2):365-378. doi: 10.1007/s00018-019-03186-2. Epub 2019 Jun 28. PMID:31254042; PMCID: PMC6971004.
Mózner O, Bartos Z, Zámbó B, Homolya L, Hegedűs T, Sarkadi B. Cellular Processing of the ABCG2 Transporter-Potential Effects on Gout and Drug Metabolism. Cells. 2019 Oct 8;8(10):1215. doi: 10.3390/cells8101215. PMID: 31597297; PMCID: PMC6830335.
Oláh A, Ruppert M, Orbán TI, Apáti Á, Sarkadi B, Merkely B, Radovits T. Hemodynamic characterization of a transgenic rat strain stably expressing the calcium sensor protein GCaMP2. Am J Physiol Heart Circ Physiol. 2019 May 1;316(5):H1224-H1228. doi: 10.1152/ajpheart.00074.2019. Epub 2019 Mar 15. PMID: 30875251.
Apáti Á, Varga N, Berecz T, Erdei Z, Homolya L, Sarkadi B. Application of human pluripotent stem cells and pluripotent stem cell-derived cellular models for assessing drug toxicity. Expert Opin Drug Metab Toxicol. 2019 Jan;15(1):61-75. doi: 10.1080/17425255.2019.1558207. Epub 2018 Dec 17. PMID:30526128.
Zámbó B, Bartos Z, Mózner O, Szabó E, Várady G, Poór G, Pálinkás M, Andrikovics H, Hegedűs T, Homolya L, Sarkadi B. Clinically relevant mutations in the ABCG2 transporter uncovered by genetic analysis linked to erythrocyte membrane protein expression. Sci Rep. 2018 May 10;8(1):7487. doi:10.1038/s41598-018-25695-z. PMID: 29749379; PMCID: PMC5945641.
Erdei Z, Schamberger A, Török G, Szebényi K, Várady G, Orbán TI, Homolya L, Sarkadi B, Apáti Á. Generation of multidrug resistant human tissues by overexpression of the ABCG2 multidrug transporter in embryonic stem cells. PLoS One. 2018 Apr 12;13(4):e0194925. doi: 10.1371/journal.pone.0194925. PMID: 29649238; PMCID: PMC5896897.
Szabó E, Türk D, Telbisz Á, Kucsma N, Horváth T, Szakács G, Homolya L, Sarkadi B, Várady G. A new fluorescent dye accumulation assay for parallel measurements of the ABCG2, ABCB1 and ABCC1 multidrug transporter functions. PLoS One. 2018 Jan 17;13(1):e0190629. doi: 10.1371/journal.pone.0190629. PMID: 29342177; PMCID: PMC5771559.
Szabó Z, Héja L, Szalay G, Kékesi O, Füredi A, Szebényi K, Dobolyi Á, Orbán TI, Kolacsek O, Tompa T, Miskolczy Z, Biczók L, Rózsa B, Sarkadi B, Kardos J. Extensive astrocyte synchronization advances neuronal coupling in slow wave activity in vivo. Sci Rep. 2017 Jul 20;7(1):6018. doi: 10.1038/s41598-017-06073-7. PMID: 28729692; PMCID: PMC5519671.
Zámbó B, Várady G, Padányi R, Szabó E, Németh A, Langó T, Enyedi Á, Sarkadi B. Decreased calcium pump expression in human erythrocytes is connected to a minor haplotype in the ATP2B4 gene. Cell Calcium. 2017 Jul;65:73-79. doi: 10.1016/j.ceca.2017.02.001. Epub 2017 Feb 3. PMID: 28216081.
Apáti Á, Berecz T, Sarkadi B. Calcium signaling in human pluripotent stem cells. Cell Calcium. 2016 Mar;59(2-3):117-23. doi: 10.1016/j.ceca.2016.01.005. Epub 2016 Feb 17. PMID: 26922096.
Apáti Á, Szebényi K, Erdei Z, Várady G, Orbán TI, Sarkadi B. The importance of drug transporters in human pluripotent stem cells and in early tissue differentiation. Expert Opin Drug Metab Toxicol. 2016;12(1):77-92. doi: 10.1517/17425255.2016.1121382. Epub 2015 Dec 14. PMID: 26592535.
Szebényi K, Füredi A, Kolacsek O, Csohány R, Prókai Á, Kis-Petik K, Szabó A, Bősze Z, Bender B, Tóvári J, Enyedi Á, Orbán TI, Apáti Á, Sarkadi B. Visualization of Calcium Dynamics in Kidney Proximal Tubules. J Am Soc Nephrol. 2015 Nov;26(11):2731-40. doi: 10.1681/ASN.2014070705. Epub 2015 Mar 18. PMID:25788535; PMCID: PMC4625667.
Várady G, Szabó E, Fehér Á, Németh A, Zámbó B, Pákáski M, Janka Z, Sarkadi B. Alterations of membrane protein expression in red blood cells of Alzheimer’s disease patients. Alzheimers Dement (Amst). 2015 Jul 21;1(3):334-8. doi: 10.1016/j.dadm.2015.06.007. PMID: 27239515; PMCID: PMC4878320.
Hegedüs C, Telbisz Á, Hegedűs T, Sarkadi B, Özvegy-Laczka C. Lipid regulation of the ABCB1 and ABCG2 multidrug transporters. Adv Cancer Res. 2015;125:97-137. doi: 10.1016/bs.acr.2014.10.004. Epub 2015 Jan 8. PMID: 25640268.
Együttműködések
Nemzetközi együttműködések
- Max Delbrück Center for Molecular Medicine, Berlin, Németország
- Paul Ehrlich Institute, Langen, Németország
- Université Paris-Saclay, Gif sur Yvette, Franciaország
- University of Lyon, IBCP, Lyon, Franciaország
- Ruđer Bošković Institute, Zagreb, Horvátország
- University of Zürich, Zürich, Svájc
- Indian Institute of Science, Bangalore, India
- NIH, Bethesda, USA
- Columbia University, New York, USA
Hazai együttműködések
- HUN-REN TTK Szerves Kémiai Intézet
- HUN-REN SZBK Biokémiai Intézet
- ELTE TTK Genetikai Tanszék
- ELTE TTK Embertani Tanszék
- Semmelweis Egyetem, Biofizikai és Sugárbiológiai Intézet
- Óbudai Egyetem
- Magyar Agrár- és Élettudományi Egyetem, Gödöllő
- Richter Gedeon Nyrt.
- Toxi-Coop Zrt.
- Gene Design Kft.
- Biospirál 2006 Kft.
- ImmunoGenes-ABS Zrt.
- Akadimpex Zrt.
- SOLVO/Charles River Company
- CellPharma Zrt.
- CelluVir Zrt.